決定木分析
概要
決定木分析では、分析の基準になる変数が最も偏る切り口でグループ分けをし、以降、各グループに対して同じ操作を繰り返すことで、予測モデルを構築します。目的変数は質的データだけでなく、量的データでも同様に分析できます。目的変数が量的データの場合は、回帰木と呼ばれます。
主な適用場面として、会員になりやすい顧客の特徴を調べる、離脱しやすいページを特定するなどが挙げられます。さらに、この決定木の概念は、ランダムフォレストとして機械学習に応用されています。
(例)どのような属性の人が会員になりやすいか
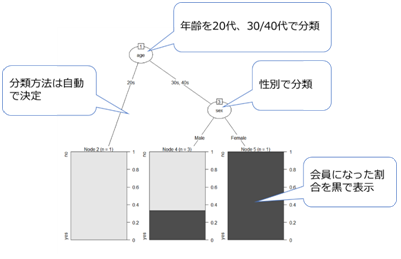
上記結果より以下がわかります。
- 20代は誰も会員にならなかった
- 30代、40代の男性は30%が会員になった
- 30代、40代の女性はすべてが会員になった
決定木分析の実行
決定木分析をするためにパッケージrpart、図の描画のためにパッケージpartykitを使います。また、データはRに組み込みのデータセットirisを使います。
ここでは、irisデータセットのSpeciesを予測するモデルを作成します。また、本分析手法はランダムに並べたりする箇所があるため、実際にコードを動かしてみた結果とここに書かれている結果とやや異なるところがでくるかもしれません。
step.1 準備
# ライブラリの読み込み
library(rpart)
library(partykit)
# データの確認
head(iris) ## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosastep.2 モデルの作成
決定木分析をするためにrpart()関数を使います。1つ目の引数で「~」の左側に目的変数(分類対象になる変数)になる列名、右側に説明変数(分類ルールの元になる変数)になる列名を指定します。次の例のようにピリオド「.」は目的変数の列を除く、すべての列を意味します。2つ目の引数でデータセットを指定します。
iris.rp <- rpart(Species~ ., data = iris)step.3 分析結果の出力
分析結果はrpart()の結果を入れたオブジェクト名をコマンドすることで得られます。*が付いている分岐は統計的に有意な分岐を意味します。
iris.rp## n= 150
##
## node), split, n, loss, yval, (yprob)
## * denotes terminal node
##
## 1) root 150 100 setosa (0.33333333 0.33333333 0.33333333)
## 2) Petal.Length< 2.45 50 0 setosa (1.00000000 0.00000000 0.00000000) *
## 3) Petal.Length>=2.45 100 50 versicolor (0.00000000 0.50000000 0.50000000)
## 6) Petal.Width< 1.75 54 5 versicolor (0.00000000 0.90740741 0.09259259) *
## 7) Petal.Width>=1.75 46 1 virginica (0.00000000 0.02173913 0.97826087) *step.4 決定木モデルの描画
モデルの描画のためにplot()関数を使います。gp 以下は文字化け対策です。 Windowsでは“Meiryo”等の日本語用フォントを指定してください。
plot(as.party(iris.rp),tp_args=T, gp = gpar(fontfamily = "Osaka", fontsize = 10))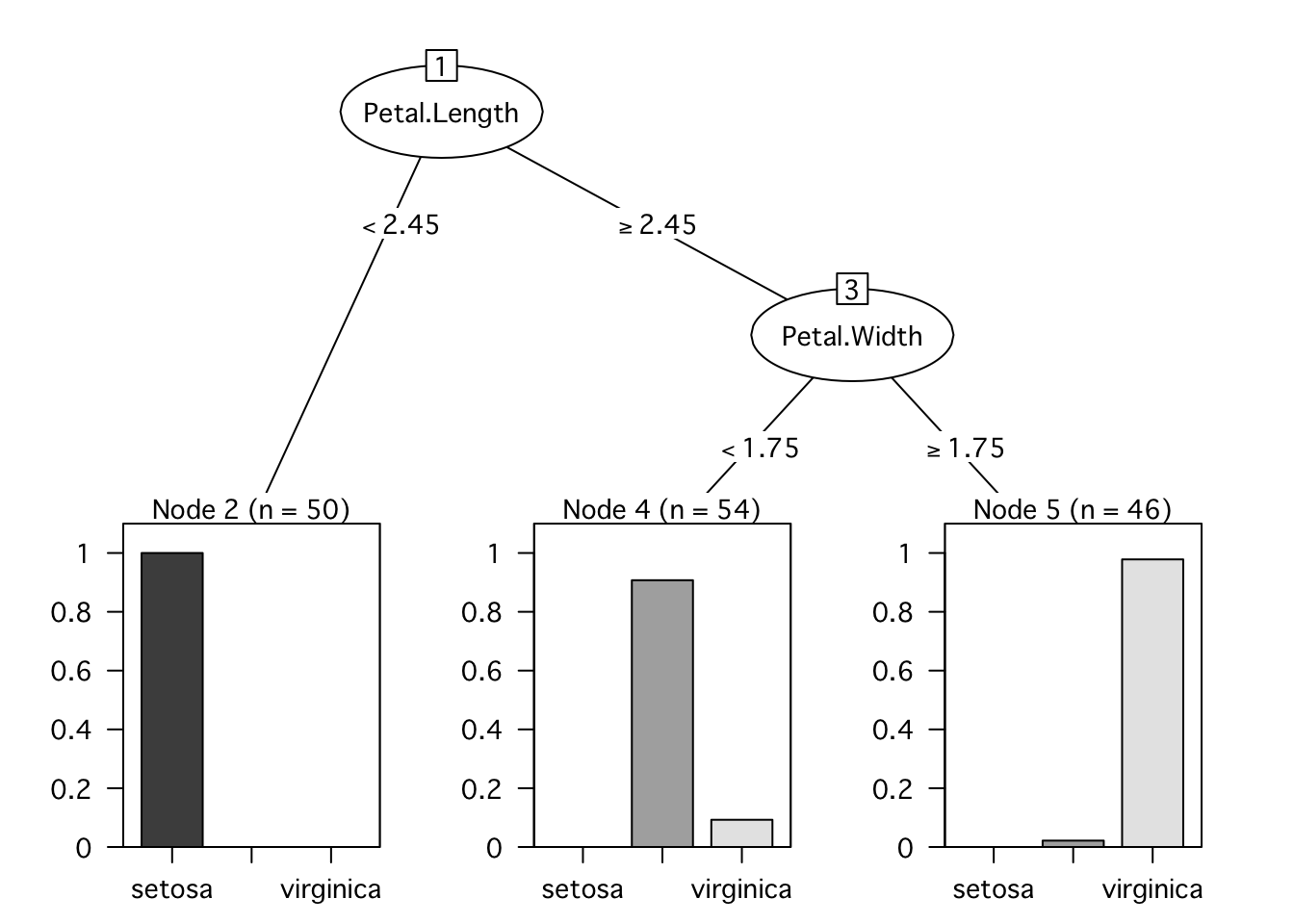
step.5 チューニング
枝の細かさ(cp)を決めるために、printcp()関数を使います。cpは枝の細かさ(深さ)を示し、値が小さくなるほど細かい枝になります。具体的には、 (xerrorの最小値 + そのxstd)より小さいxerrorのうち最大値になるもののcpを木の剪定に用います(あくまで目安です)。
printcp(iris.rp)##
## Classification tree:
## rpart(formula = Species ~ ., data = iris)
##
## Variables actually used in tree construction:
## [1] Petal.Length Petal.Width
##
## Root node error: 100/150 = 0.66667
##
## n= 150
##
## CP nsplit rel error xerror xstd
## 1 0.50 0 1.00 1.16 0.051277
## 2 0.44 1 0.50 0.68 0.060970
## 3 0.01 2 0.06 0.10 0.030551plotcp(iris.rp)
step.6 再分析
先ほどチューニングで得られたcpを、rpart()関数中で指定します。
iris.rp <- rpart(Species~ ., data=iris, cp = 0.01)